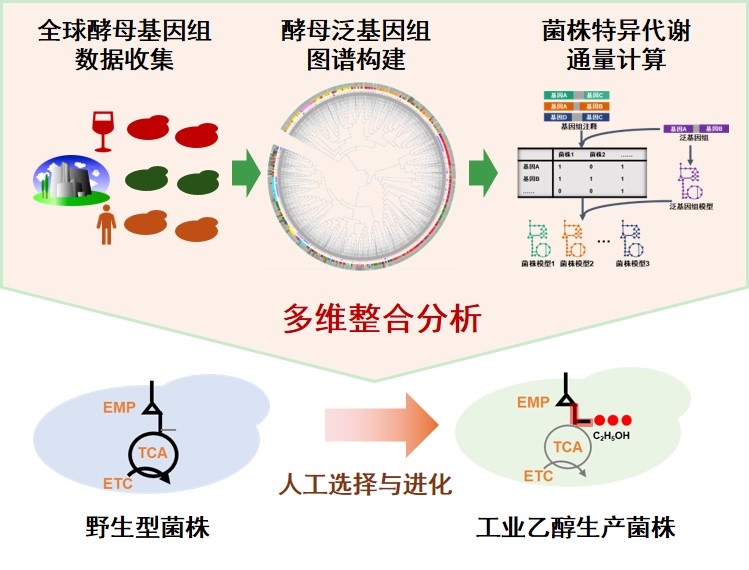
近日,大連化學物理研究所生物技術研究部合成微生物學研究組(1823組)周雍進研究員團隊與上海交通大學魯洪中副教授合作,在酵母系統生物學研究中取得新進展。研究團隊通過整合分析全球1807株釀酒酵母菌株的基因組與生態位數據,構建了高覆蓋度的釀酒酵母泛基因組及菌株特異性代謝模型,系統解析了酵母通過基因組精簡與代謝重編程適應多樣化環境的分子機制。
釀酒酵母在烘焙、釀酒等領域具有悠久的應用歷史,在自然與人工環境中均展現出強大的適應性進化能力。盡管不同進化分支的菌株在遺傳和表型上存在顯著多樣性,但其在代謝水平上的差異尚不清晰。目前,全球科研與工業界主要依賴少數實驗室菌株(如S288c,CEN.PK)作為出發菌株,限制了對酵母寶貴自然多樣性資源的開發與利用。因此,如何篩選最優菌株以構建服務于醫藥、能源、化工等領域的酵母細胞工廠,成為亟待解決的問題。
本研究創建了高質量酵母泛基因組與菌株特異性代謝模型數字資源庫。基于此,團隊建立了“泛基因組-代謝模型-多組學整合”的多維度分析流程,為從系統層面理解微生物的環境適應性及代謝特性提供了新理論框架。以工業乙醇生產菌株為例,團隊從基因、轉錄及代謝通量三個維度證實,強化糖酵解下游途徑與乙醇的高效合成密切相關,該發現為后續乙醇及其衍生物細胞工廠的理性設計提供了重要線索。
該工作以“Yeast adapts to diverse ecological niches driven by genomics and metabolic reprogramming”為題,發表在《美國國家科學院院刊》(Proceedings of the National Academy of Sciences)上。我所1823組博士研究生王浩宇為論文第一作者,丹麥生物創新研究院Jens Nielsen教授為文章提供了重要指導。以上工作得到國家重點研發計劃、國家自然科學基金等項目的資助。(文/圖 王浩宇)